1、骨架提取
骨架提取,也叫二值图像细化。这种算法能将一个连通区域细化成一个像素的宽度,用于特征提取和目标拓扑表示。
morphology子模块提供了两个函数用于骨架提取,分别是Skeletonize()函数和medial_axis()函数。
1.1. Skeletonize()函数
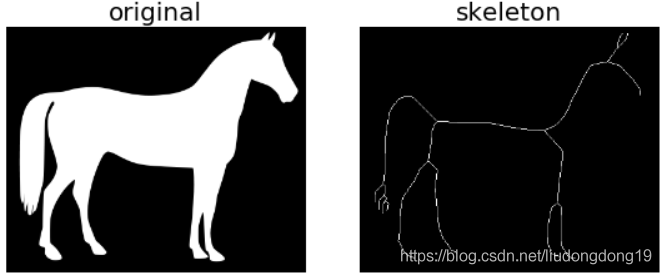
输入和输出都是一幅二值图像。
from skimage import morphology,data,color
import matplotlib.pyplot as plt
image=color.rgb2gray(data.horse())
image=1-image #反相
#实施骨架算法
skeleton =morphology.skeletonize(image)
#显示结果
fig, (ax1, ax2) = plt.subplots(nrows=1, ncols=2, figsize=(8, 4))
ax1.imshow(image, cmap=plt.cm.gray)
ax1.axis('off')
ax1.set_title('original', fontsize=20)
ax2.imshow(skeleton, cmap=plt.cm.gray)
ax2.axis('off')
ax2.set_title('skeleton', fontsize=20)
fig.tight_layout()
plt.show()
1.2. medial_axis(img,mask=None,return_distance=False)
mask: 掩模。默认为None, 如果给定一个掩模,则在掩模内的像素值才执行骨架算法。
return_distance: bool型值,默认为False. 如果为True, 则除了返回骨架,还将距离变换值也同时返回。这里的距离指的是中轴线上的所有点与背景点的距离。
import numpy as npimport scipy.ndimage as ndifrom skimage import morphologyimport matplotlib.pyplot as plt #编写一个函数,生成测试图像def microstructure(l=256): n = 5 x, y = np.ogrid[0:l, 0:l] mask = np.zeros((l, l)) generator = np.random.RandomState(1) points = l * generator.rand(2, n**2) mask[(points[0]).astype(np.int), (points[1]).astype(np.int)] = 1 mask = ndi.gaussian_filter(mask, sigma=l/(4.*n)) return mask > mask.mean() data = microstructure(l=64) #生成测试图像 #计算中轴和距离变换值skel, distance =morphology.medial_axis(data, return_distance=True) #中轴上的点到背景像素点的距离dist_on_skel = distance * skel fig, (ax1, ax2) = plt.subplots(1, 2, figsize=(8, 4))ax1.imshow(data, cmap=plt.cm.gray, interpolation='nearest')#用光谱色显示中轴ax2.imshow(dist_on_skel, cmap=plt.cm.spectral, interpolation='nearest')ax2.contour(data, [0.5], colors='w') #显示轮廓线 fig.tight_layout()plt.show()
2. 分水岭算法
分水岭算法是一种用于图像分割的经典算法,是基于拓扑理论的数学形态学的分割方法。如果图像中的目标物体是连在一起的,则分割起来会更困难,分水岭算法经常用于处理这类问题,通常会取得比较好的效果。
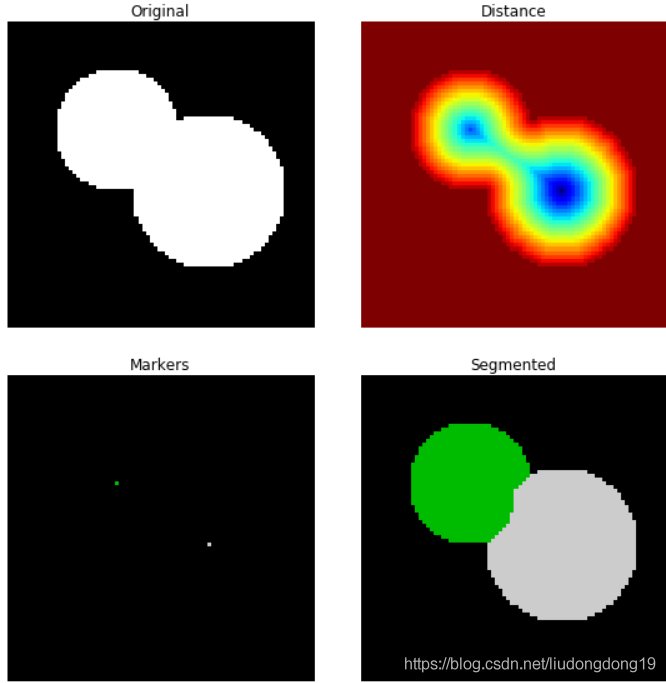
2.1. 基于距离变换的分山岭图像分割
import numpy as npimport matplotlib.pyplot as pltfrom scipy import ndimage as ndifrom skimage import morphology,feature #创建两个带有重叠圆的图像x, y = np.indices((80, 80))x1, y1, x2, y2 = 28, 28, 44, 52r1, r2 = 16, 20mask_circle1 = (x - x1)**2 + (y - y1)**2 < r1**2mask_circle2 = (x - x2)**2 + (y - y2)**2 < r2**2image = np.logical_or(mask_circle1, mask_circle2) #现在我们用分水岭算法分离两个圆distance = ndi.distance_transform_edt(image) #距离变换local_maxi =feature.peak_local_max(distance, indices=False, footprint=np.ones((3, 3)), labels=image) #寻找峰值markers = ndi.label(local_maxi)[0] #初始标记点labels =morphology.watershed(-distance, markers, mask=image) #基于距离变换的分水岭算法 fig, axes = plt.subplots(nrows=2, ncols=2, figsize=(8, 8))axes = axes.ravel()ax0, ax1, ax2, ax3 = axes ax0.imshow(image, cmap=plt.cm.gray, interpolation='nearest')ax0.set_title("Original")ax1.imshow(-distance, cmap=plt.cm.jet, interpolation='nearest')ax1.set_title("Distance")ax2.imshow(markers, cmap=plt.cm.spectral, interpolation='nearest')ax2.set_title("Markers")ax3.imshow(labels, cmap=plt.cm.spectral, interpolation='nearest')ax3.set_title("Segmented") for ax in axes: ax.axis('off') fig.tight_layout()plt.show()
2.2. 基于梯度的分水岭图像分割
import matplotlib.pyplot as pltfrom scipy import ndimage as ndifrom skimage import morphology,color,data,filter image =color.rgb2gray(data.camera())denoised = filter.rank.median(image, morphology.disk(2)) #过滤噪声 #将梯度值低于10的作为开始标记点markers = filter.rank.gradient(denoised, morphology.disk(5)) <10markers = ndi.label(markers)[0] gradient = filter.rank.gradient(denoised, morphology.disk(2)) #计算梯度labels =morphology.watershed(gradient, markers, mask=image) #基于梯度的分水岭算法 fig, axes = plt.subplots(nrows=2, ncols=2, figsize=(6, 6))axes = axes.ravel()ax0, ax1, ax2, ax3 = axes ax0.imshow(image, cmap=plt.cm.gray, interpolation='nearest')ax0.set_title("Original")ax1.imshow(gradient, cmap=plt.cm.spectral, interpolation='nearest')ax1.set_title("Gradient")ax2.imshow(markers, cmap=plt.cm.spectral, interpolation='nearest')ax2.set_title("Markers")ax3.imshow(labels, cmap=plt.cm.spectral, interpolation='nearest')ax3.set_title("Segmented") for ax in axes: ax.axis('off') fig.tight_layout()plt.show()







评论(0)
您还未登录,请登录后发表或查看评论